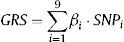En estudios recientes se han identificado varias variantes genéticas asociadas a la enfermedad coronaria. Algunas de estas variantes genéticas no se asocian a factores de riesgo cardiovascular clásicos y no están claros los mecanismos por los que se producen tales asociaciones. El objetivo de este estudio es determinar si estas variantes genéticas están relacionadas con la aterosclerosis subclínica medida con el grosor intimomedial carotídeo, la rigidez carotídea y el índice tobillo-brazo.
MétodosSe llevó a cabo un estudio transversal anidado en el seguimiento de la cohorte REGICOR. El estudio se llevó a cabo en 2.667 individuos. Se realizaron mediciones de la aterosclerosis subclínica con métodos estandarizados. Se determinaron los genotipos relativos a nueve variantes genéticas para evaluar las asociaciones con la aterosclerosis subclínica, individualmente y con una puntuación de riesgo genético ponderada. Se llevó a cabo una revisión sistemática y metanálisis de los estudios previos que analizaron esas asociaciones.
ResultadosNinguna de las variantes genéticas estudiadas ni la puntuación de riesgo genético mostraron una asociación significativa con la aterosclerosis subclínica. En el metanálisis, el alelo de riesgo del rs1746048 (CXCL12) (n = 10.581) mostró asociación directa con el grosor intimomedial carotídeo (β = 0,008; intervalo de confianza del 95%, 0,001-0,015), mientras que el alelo de riesgo rs6725887 (WDR12) (n = 7.801) mostró asociación inversa (β = –0,013; intervalo de confianza del 95%, –0,024 a –0,003).
ConclusionesLas variantes genéticas analizadas parecen intervenir en la asociación con la enfermedad coronaria por diferentes mecanismos. Estos resultados generan la hipótesis de que la variante CXCL12 parece influir en el riesgo de enfermedad coronaria a través del remodelado y el engrosamiento de las arterias, mientras que la variante de riesgo WDR12 podría estar relacionada con una mayor vulnerabilidad de la placa.
Palabras clave
La enfermedad coronaria (EC), que es la principal causa mundial de muerte1 y discapacidad2, tiene un importante componente genético. En estudios recientes de asociación de genoma completo, se han identificado varios polimorfismos de un solo nucleótido (SNP) asociados al infarto de miocardio y la EC3–11. Algunas de estas variantes se asocian también a los factores de riesgo cardiovascular clásicos (FRCVc), pero esto no ocurre en otras y los mecanismos biológicos que subyacen a su asociación con la EC siguen sin estar claros.
La aterosclerosis es el principal mecanismo etiopatogénico asociado a la EC. Empieza con una fase subclínica, que se inicia en una etapa temprana de la vida12, y sus manifestaciones clínicas constituyen el estadio final de este proceso crónico. En consecuencia, la medición de las etapas iniciales e intermedias de la aterosclerosis subclínica puede utilizarse para identificar la progresión de este proceso y predecir el riesgo futuro de presentar episodios cardiovasculares o coronarios. El grosor intimomedial (GIM), la rigidez de la arteria carótida y el índice tobillo-brazo (ITB) son marcadores de la aterosclerosis exactos, cuantificables, reproducibles y no invasivos y constituyen factores predictivos claramente establecidos de los eventos cardiovasculares futuros, como el infarto de miocardio y el ictus13–15.
El estudio de la asociación entre la presencia de aterosclerosis subclínica y la de los SNP asociados a la EC de manera independiente de los FRCVc debería aportar cierta luz sobre los mecanismos biológicos que subyacen a esta relación. Este estudio tiene dos objetivos: a) determinar en un estudio de base poblacional si las variantes genéticas asociadas a la EC pero no a los FRCVc están relacionadas con la aterosclerosis subclínica evaluada mediante el GIM carotídeo, la rigidez carotídea y el ITB, y b) llevar a cabo una revisión sistemática actualizada y un metanálisis de los estudios previos que han analizado estas asociaciones.
MÉTODOSDiseñoSe llevó a cabo un estudio transversal anidado en el estudio de cohorte Registre Gironí del Cor (REGICOR)16,17. Se utilizaron los datos del seguimiento de dos cohortes de base poblacional, creadas inicialmente en los años 1995 y 2000 (con tasas de respuesta del 72,4 y el 70,0% respectivamente) en ciudades representativas de la diversidad geográfica de la provincia de Girona16,17. Durante el periodo 2007-2010, se invitó a los participantes supervivientes no ingresados que residían en esas localidades a acudir a una visita de seguimiento; la tasa de respuesta fue del 78,4%. Se seleccionó a los participantes de entre 35 y 74 años de edad que el momento de los exámenes basales no presentaban enfermedad cardiovascular en ese momento y de cuyo ADN se disponía. El comité ético local aprobó este estudio y los participantes dieron su consentimiento informado por escrito.
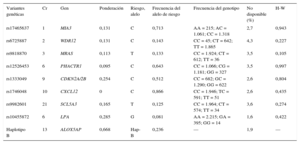
Selección de las variantes genéticas, determinación del genotipo y generación de la puntuación de riesgo multilocusSe seleccionaron nueve variantes genéticas asociadas a la EC pero no asociadas a los FRCVc: presión arterial, colesterol total, colesterol unido a lipoproteínas de baja densidad, colesterol unido a lipoproteínas de alta densidad, triglicéridos, diabetes mellitus y tabaquismo. Se generó una puntuación de riesgo genético multilocus de la forma descrita con anterioridad17. Las primeras siete variantes seleccionadas se asociaron a la EC en estudios de asociación de genoma completo: rs17465637 en MIA3, rs6725887 en WDR12, rs9818870 en MRAS, rs12526453 en PHACTR1, rs1333049 cerca de CDKN2A/2B, rs1746048 cerca de CXCL12 y rs9982601 cerca de SCL5A3. Se incluyó también la variante rs10455872 en LPA18 y el haplotipo B (rs10507391, rs17216473, rs9315050, rs17222842) en ALOX5AP19, que se han asociado al riesgo de EC de manera independiente de los FRCVc.
El Centro Nacional de Investigaciones Oncológicas, con base en Madrid, determinó el genotipo en las muestras de ADN usando el chip Cardio inCode (Ferrer inCode; Barcelona, España), que tiene su base en Veracode (Illumina; San Diego, Estados Unidos) y tecnologías KASPar (KBioscience; Hoddesdon, Reino Unido). Se aplicaron criterios de control de calidad tanto a cada una de las muestras como a los SNP seleccionados, incluidos los análisis de equilibrio de Hardy-Weinberg. La coincidencia global del chip con la tecnología de referencia fue del 99,9% y la sensibilidad y la especificidad del método analítico fueron superiores al 98,6%.
Se calculó también una puntuación de riesgo genético multilocus para cada individuo mediante la suma del número de alelos de riesgo en las nueve variantes, tras ponderar cada variante con la magnitud estimada de su efecto en el estudio CARDIoGRAM10 (tabla 1). Las ponderaciones de las variantes de LPA y ALOX5AP se basaron en los coeficientes descritos en las correspondientes publicaciones18,19. Se calculó la puntuación de riesgo genético empleando la siguiente fórmula:
Donde: βi es la magnitud estimada del efecto descrito para cada variante en el estudio CARDIoGRAM o la correspondiente publicación para LPA y ALOX5AP, y SNPi es el número de alelos de riesgo de cada uno de los SNP evaluados (que puede adoptar valores de 0, 1 o 2 para los SNP genotipificados).Algunas variantes genéticas seleccionadas e incluidas en este estudio, ponderaciones asignadas a cada variante, frecuencias de alelos observados y genotipos, datos no disponibles y valor de p para la desviación respecto al equilibrio de Hardy-Weinberg
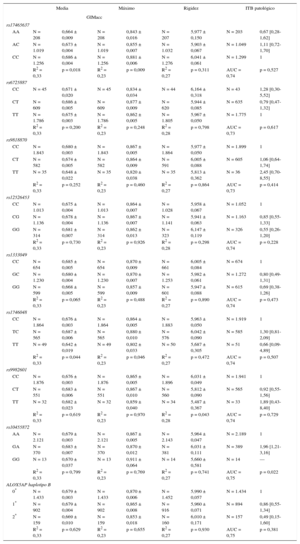
| Variantes genéticas | Cr | Gen | Ponderación | Riesgo, alelo | Frecuencia del alelo de riesgo | Frecuencia del genotipo | No disponible (%) | H-W |
|---|---|---|---|---|---|---|---|---|
| rs17465637 | 1 | MIA3 | 0,131 | C | 0,713 | AA = 215; AC = 1.061; CC = 1.318 | 2,7 | 0,943 |
| rs6725887 | 2 | WDR12 | 0,131 | C | 0,143 | CC = 45; CT = 642; TT = 1.865 | 4,3 | 0,227 |
| rs9818870 | 3 | MRAS | 0,113 | T | 0,133 | CC = 1.924; CT = 612; TT = 36 | 3,5 | 0,105 |
| rs12526453 | 6 | PHACTR1 | 0,095 | C | 0,643 | CC = 1.066; CG = 1.181; GG = 327 | 3,5 | 0,997 |
| rs1333049 | 9 | CDKN2A/2B | 0,254 | C | 0,512 | CC = 682; GC = 1.290; GG = 622 | 2,6 | 0,804 |
| rs1746048 | 10 | CXCL12 | 0 | C | 0,866 | CC = 1.946; TC = 591; TT = 51 | 2,6 | 0,435 |
| rs9982601 | 21 | SCL5A3 | 0,165 | T | 0,125 | CC = 1.964; CT = 574; TT = 34 | 3,6 | 0,274 |
| rs10455872 | 6 | LPA | 0,285 | G | 0,081 | AA = 2.215; GA = 395; GG = 14 | 1,6 | 0,422 |
| Haplotipo B | 13 | ALOX5AP | 0,668 | Hap-B | 0,236 | — | 1,9 | — |
Tres ecografistas titulados evaluaron las exploraciones carotídeas usando un protocolo estandarizado y un equipo de ecografía Acuson XP128 (Acuson-Siemens; Erlangen, Alemania), equipado con un transductor de haz lineal de L7 5–12MHz. De forma resumida, se examinaron los siguientes segmentos arteriales predefinidos de las arterias coronarias derecha e izquierda: a) la arteria carótida común en una posición 1cm proximal a la dilatación; b) el bulbo carotídeo, y c) la carótida interna en una posición 1cm distal a la división del flujo. Se registró una imagen estática de cada segmento arterial como imagen estática DICOM (digital information and communication in medicine). Los archivos de imagen se transfirieron a través de un sistema seguro a un laboratorio de interpretación (Imagelabonline; Ámsterdam, Países Bajos). Técnicos específicamente formados y titulados analizaron las imágenes con el programa informático validado «eTrack»20. Los técnicos identificaron las interfases con el punto de mira; el programa informático conecta el punto de mira con curvas spline. El GIM carotídeo se definió como la distancia media (GIM medio) y la distancia máxima (GIM máximo) entre las interfases luz-íntima y media-adventicia en determinado segmento de la pared arterial lejana.
Los resultados de interés fueron: a) el promedio de los GIM medios izquierdo y derecho de las carótidas comunes (GIMacc medio), las arterias carótidas internas (GIMaci medio) y los bulbos carotídeos (GIMbul medio), y b) el valor máximo del GIM de los lados izquierdo y derecho de las arterias carótidas comunes (GIMacc máximo), las arterias carótidas internas (GIMaci máximo) y los bulbos carotídeos (GIMbul máximo).
Se evaluó la variabilidad interecografistas e intraecografista empleando mediciones repetidas del GIM realizadas a 42 participantes en dos visitas separadas 2 semanas por hasta tres ecografistas. El estudio de la repetibilidad mostró unos coeficientes de correlación intraclase > 0,82 para el GIMacc medio, tanto para la variabilidad intraecografista como para la interecografistas.
Rigidez carotídeaPara la evaluación de la rigidez carotídea, se obtuvo el coeficiente de distensibilidad arterial (CD), definido como el cambio relativo del área de corte transversal por unidad de presión. Durante el examen de ecografía carotídea, se visualizaron en modo B las paredes anterior y posterior de las arterias carótidas comunes derecha e izquierda en la parte distal. Para obtener el registro en modo M de íntima-luz de la pared anterior y de luz-íntima de la pared posterior, el ecografista pasó del modo B a una imagen de 1/3 modo B y 2/3 modo M de la carótida común en la parte distal. La imagen de 1/3 modo B guía la de modo M. El movimiento de las paredes arteriales en la imagen de 2/3 modo M muestra las formas de onda con los patrones de doble línea de las paredes arteriales a lo largo del tiempo. El análisis de las imágenes se llevó a cabo con el programa informático validado «eTrack». El programa establece un trazado de las formas de onda de los bordes de avance de las interfases de íntima-luz de la pared anterior y de luz-íntima de la pared posterior. Si se identifican los contornos de ambas paredes durante al menos dos latidos cardiacos, el programa permite calcular los parámetros del diámetro de la luz y las frecuencias cardiacas. Con base en esta información, se puede obtener otros parámetros de valoración (p. ej., el CD). Se estimó el cambio del diámetro durante el ciclo cardiaco o la distensión (ΔD), y se calculó el CD como sigue21:
Donde: CD es el coeficiente de distensibilidad, ΔD es la distensión, D es el diámetro diastólico y ΔP es la presión del pulso (la presión arterial se determinó inmediatamente antes del examen ecográfico).Se promediaron los valores de CD de las carótidas izquierda y derecha. Si faltaban los datos de modo M de una localización, se utilizaban únicamente los datos disponibles de la otra.
Índice tobillo-brazoSe determinó la presión arterial sistólica en decúbito, después de 5min en reposo, en la arteria humeral del brazo derecho y las arterias tibial posterior y pedia dorsal de ambas piernas, utilizando un aparato Doppler continuo. Se calculó el ITB derecho e izquierdo mediante el cociente del valor más alto de la presión de la pierna respecto a la presión del brazo, y para el análisis el valor más bajo de los dos cocientes ITB se clasificó como bajo (< 0,9), normal (0,9–1,3) o alto (> 1,3)16.
Otras variablesRealizó los exámenes un equipo de enfermeras y entrevistadores adecuadamente capacitados. Se utilizó una balanza de precisión y de calibrado sencillo para las mediciones de peso y talla, con los participantes en ropa interior y descalzos; se determinó el índice de masa corporal dividiendo el peso en kilos por el cuadrado de la estatura en metros. La presión arterial se determinó con un esfigmomanómetro (OMRON 711) calibrado periódicamente. Se utilizó un manguito apropiado para el tamaño de la parte superior del brazo en cada participante. Las mediciones se hicieron con el paciente sentado tras un reposo de 5min. Se efectuaron dos lecturas y se registró para el estudio el valor más bajo.
Se utilizaron cuestionarios estandarizados para obtener las variables sociodemográficas y de estilo de vida, así como para los antecedentes previos y los tratamientos de la diabetes mellitus, la hipertensión y la hipercolesterolemia. El tabaquismo actual se definió como un tabaquismo activo durante el año anterior.
Se extrajeron muestras de sangre después de 10–14h en ayunas. Las concentraciones de colesterol total, colesterol unido a lipoproteínas de alta densidad y triglicéridos se determinaron con métodos directos (Roche Diagnostics; Basilea, Suiza).
Análisis estadísticoLas variables continuas se resumieron mediante la media ± desviación estándar o la mediana [intervalo intercuartílico] cuando la distribución se apartaba de la normal, y las variables discretas se presentaron como proporciones.
Para estimar la asociación existente entre las diferentes variantes genéticas y las mediciones de la aterosclerosis subclínica, se utilizaron modelos lineales generales mixtos ajustados por edad y sexo y regresión logística, tomando la encuesta como variable aleatoria.
Se estableció la fase de ALOX5AP haplotipo B empleando los genotipos de los cuatro SNP que definen el haplotipo y la presencia de los alelos AAAG en los SNP rs10507391, rs17216473, rs9315050 y rs17222842 respectivamente. La determinación de la fase se realizó con la función haplo.em del paquete haplo.stats22, y se obtuvo la probabilidad de que hubiera 0, 1 o 2 copias del haplotipo de riesgo en cada muestra individual. Esta probabilidad se añadió a la puntuación de riesgo genético, como con los ocho SNP. El análisis estadístico se llevó a cabo con el programa informático R (versión 3.0.1)23. Para tener en cuenta la realización de comparaciones múltiples, se estableció un valor de p con corrección de Bonferroni < 0,006 (0,05/9 variantes genéticas independientes = 0,006) para definir como estadísticamente significativa una diferencia.
Revisión sistemática y metanálisisRevisión de la bibliografíaSe siguieron las recomendaciones de la declaración PRISMA para la mejora de la publicación de revisiones sistemáticas y metanálisis24. Se definió un protocolo de revisión interna, básicamente con una búsqueda detallada en la base de datos PubMed desde su inicio hasta julio de 2013, para identificar los estudios que evaluaban la relación entre las variantes genéticas de interés y la aterosclerosis subclínica. La interrogación a la base de datos se hizo con los siguientes términos de búsqueda: («subclinical atherosclerosis» OR «carotid» OR «thickness» OR «stiffness» OR «plaque» OR «ankle-brachial index») y para cada variante genética se añadió también («rs id» OR «gene name»). No se impusieron limitaciones de idioma a priori.
Dos revisores independientes (MZ y RE) examinaron manualmente para su selección los artículos identificados mediante esta búsqueda, primero basándose en el título, luego en el resumen y finalmente en el manuscrito completo, para evaluar su idoneidad para la inclusión en el metanálisis. Se examinaron también las referencias bibliográficas citadas en esos artículos para identificar las cifras relevantes no detectadas en la búsqueda inicial.
Extracción de los datosDos investigadores (MZ y RE) extrajeron los datos de manera independiente: diseño del estudio; variables de valoración evaluadas; características de la cohorte; genotipo(s), frecuencias alélicas y modelo genético utilizado en el análisis; ajuste multivariable, y las estimaciones de las asociaciones. Las discrepancias se resolvieron por consenso.
Análisis estadísticoLos análisis estadísticos del metanálisis se realizaron con la función meta.DSL del paquete rmeta R25. Se calcularon los coeficientes de regresión lineal globales, los intervalos de confianza del 95% y los valores de p bilaterales según modelos de efectos aleatorios (DerSimonian y Laird)26. Se evaluó la heterogeneidad de los efectos entre los estudios calculando el parámetro estadístico I2.
Cuando los manuscritos definían más de un estudio o población, se consideraron para el metanálisis los resultados de los estudios individuales. Por el contrario, si un estudio estaba incluido en más de un manuscrito, solamente se tuvo en cuenta los resultados presentados en el manuscrito que indicaba un tamaño muestral más alto. Se utilizaron métodos de gráficos de embudo para evaluar la posible existencia de un sesgo de publicación. Se previeron análisis de metarregresión para explicar las posibles causas de heterogeneidad, incluida la del tamaño muestral de los estudios, la proporción de varones respecto a mujeres, la media de edad y la media de GIM de la población.
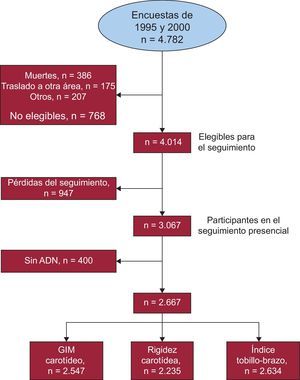
RESULTADOSDe los 4.782 individuos inicialmente incluidos en las dos encuestas, se consideró que 4.014 eran elegibles para el seguimiento, 3.067 (76,4%) acudieron al segundo examen y de 2.667 (66,4%) se dispuso de muestras de ADN (figura). En la tabla 1 se muestra la frecuencia de los alelos, la frecuencia del genotipo, la falta de genotipo disponible y el valor de p para el equilibrio de Hardy-Weinberg. La frecuencia alélica observada concordaba con lo anteriormente descrito en HapMap para la población caucásica, y las frecuencias genotípicas concordaban con el equilibrio de Hardy-Weinberg. La falta de genotipo disponible fue < 5% con todos los SNP considerados en estos análisis.
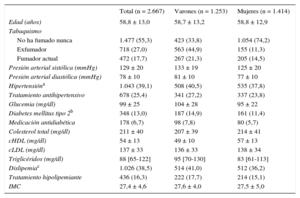
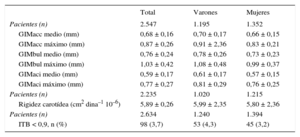
En la tabla 2 se presentan las principales características sociodemográficas y el perfil de FRCVc de los participantes en el estudio. En la tabla 3 se indican las características de las mediciones de la aterosclerosis subclínica observada en la muestra. En general, la carga aterosclerótica era mayor en los varones que en las mujeres.
Datos sociodemográficos y principales características clínicas de la muestra total y estratificados según el sexo de los participantes en este estudio
| Total (n = 2.667) | Varones (n = 1.253) | Mujeres (n = 1.414) | |
|---|---|---|---|
| Edad (años) | 58,8 ± 13,0 | 58,7 ± 13,2 | 58,8 ± 12,9 |
| Tabaquismo | |||
| No ha fumado nunca | 1.477 (55,3) | 423 (33,8) | 1.054 (74,2) |
| Exfumador | 718 (27,0) | 563 (44,9) | 155 (11,3) |
| Fumador actual | 472 (17,7) | 267 (21,3) | 205 (14,5) |
| Presión arterial sistólica (mmHg) | 129 ± 20 | 133 ± 19 | 125 ± 20 |
| Presión arterial diastólica (mmHg) | 78 ± 10 | 81 ± 10 | 77 ± 10 |
| Hipertensióna | 1.043 (39,1) | 508 (40,5) | 535 (37,8) |
| Tratamiento antihipertensivo | 678 (25,4) | 341 (27,2) | 337 (23,8) |
| Glucemia (mg/dl) | 99 ± 25 | 104 ± 28 | 95 ± 22 |
| Diabetes mellitus tipo 2b | 348 (13,0) | 187 (14,9) | 161 (11,4) |
| Medicación antidiabética | 178 (6,7) | 98 (7,8) | 80 (5,7) |
| Colesterol total (mg/dl) | 211 ± 40 | 207 ± 39 | 214 ± 41 |
| cHDL (mg/dl) | 54 ± 13 | 49 ± 10 | 57 ± 13 |
| cLDL (mg/dl) | 137 ± 33 | 136 ± 33 | 138 ± 34 |
| Triglicéridos (mg/dl) | 88 [65-122] | 95 [70-130] | 83 [61-113] |
| Dislipemiac | 1.026 (38,5) | 514 (41,0) | 512 (36,2) |
| Tratamiento hipolipemiante | 436 (16,3) | 222 (17,7) | 214 (15,1) |
| IMC | 27,4 ± 4,6 | 27,6 ± 4,0 | 27,5 ± 5,0 |
cHDL: colesterol unido a lipoproteínas de alta densidad; cLDL: colesterol unido a lipoproteínas de baja densidad; IMC: índice de masa corporal.
Los datos expresan n (%), media ± desviación estándar o mediana [intervalo intercuartílico].
Determinaciones de los parámetros carotídeos estudiados y proporción de individuos con índice tobillo-brazo patológico en el conjunto de la muestra y con estratificación por sexo
| Total | Varones | Mujeres | |
|---|---|---|---|
| Pacientes (n) | 2.547 | 1.195 | 1.352 |
| GIMacc medio (mm) | 0,68 ± 0,16 | 0,70 ± 0,17 | 0,66 ± 0,15 |
| GIMacc máximo (mm) | 0,87 ± 0,26 | 0,91 ± 2,36 | 0,83 ± 0,21 |
| GIMbul medio (mm) | 0,76 ± 0,24 | 0,78 ± 0,26 | 0,73 ± 0,23 |
| GIMbul máximo (mm) | 1,03 ± 0,42 | 1,08 ± 0,48 | 0,99 ± 0,37 |
| GIMaci medio (mm) | 0,59 ± 0,17 | 0,61 ± 0,17 | 0,57 ± 0,15 |
| GIMaci máximo (mm) | 0,77 ± 0,27 | 0,81 ± 0,29 | 0,76 ± 0,25 |
| Pacientes (n) | 2.235 | 1.020 | 1.215 |
| Rigidez carotídea (cm2 dina–1 10–6) | 5,89 ± 0,26 | 5,99 ± 2,35 | 5,80 ± 2,36 |
| Pacientes (n) | 2.634 | 1.240 | 1.394 |
| ITB < 0,9, n (%) | 98 (3,7) | 53 (4,3) | 45 (3,2) |
GIMacc: grosor intimomedial de la arteria carótida común; GIMaci: grosor intimomedial de la arteria carótida interna; GIMbul: grosor intimomedial en el bulbo carotídeo; ITB: índice tobillo-brazo.
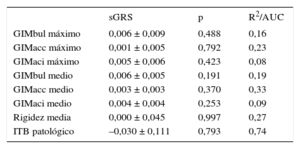
Teniendo en cuenta el tamaño muestral, se dispuso de una potencia estadística suficiente (80%) para detectar un coeficiente β > 0,040, 0,030, 0,026, 0,024 y 0,024mm para el GIM de la carótida común, considerando un nivel de significación nominal (α = 0,05), un modelo aditivo genético y una frecuencia alélica de 0,10, 0,20, 0,30, 0,40 y 0,50 respectivamente. En la tabla 4 se detalla la asociación entre los diferentes SNP y las mediciones de la aterosclerosis subclínica (GIMacc medio, GIMacc máximo, rigidez carotídea e ITB patológico). La asociación con las demás mediciones de la aterosclerosis subclínica (GIMbul medio, GIMbul máximo, GIMaci medio y GIMaci máximo) se muestran en la tabla 1 del material suplementario. Tan solo el rs10455872 (genotipo GA frente a AA) tendió a asociarse a mayor probabilidad de ITB patológico (odds ratio = 1,96; intervalo de confianza del 95%, 1,21-3,17; p = 0,022). No se observó ninguna otra asociación uniforme entre un SNP y alguna medición de la aterosclerosis. La puntuación de riesgo genético ponderada no estaba asociada con los diferentes indicadores de aterosclerosis evaluados en este estudio (tabla 5).
Análisis de los diversos grupos genotípicos con ajuste respecto a la edad y el sexo
| Media | Máximo | Rigidez | ITB patológico | |||||
|---|---|---|---|---|---|---|---|---|
| GIMacc | ||||||||
| rs17465637 | ||||||||
| AA | N = 208 | 0,664 ± 0,009 | N = 208 | 0,843 ± 0,016 | N = 207 | 5,977 ± 0,150 | N = 203 | 0,67 [0,28-1,62] |
| AC | N = 1.019 | 0,673 ± 0,004 | N = 1.019 | 0,855 ± 0,007 | N = 1.032 | 5,903 ± 0,067 | N = 1.049 | 1,11 [0,72-1,70] |
| CC | N = 1.256 | 0,686 ± 0,004 | N = 1.256 | 0,881 ± 0,006 | N = 1.276 | 6,041 ± 0,061 | N = 1.299 | 1 |
| R2 = 0,33 | p = 0,018 | R2 = 0,23 | p = 0,009 | R2 = 0,27 | p = 0,311 | AUC = 0,74 | p = 0,527 | |
| rs6725887 | ||||||||
| CC | N = 45 | 0,671 ± 0,020 | N = 45 | 0,834 ± 0,034 | N = 44 | 6,164 ± 0,318 | N = 43 | 1,28 [0,30-5,52] |
| CT | N = 609 | 0,686 ± 0,005 | N = 609 | 0,877 ± 0,009 | N = 620 | 5,944 ± 0,085 | N = 635 | 0,79 [0,47-1,32] |
| TT | N = 1.786 | 0,675 ± 0,003 | N = 1.786 | 0,862 ± 0,005 | N = 1.805 | 5,967 ± 0,050 | N = 1.775 | 1 |
| R2 = 0,33 | p = 0,200 | R2 = 0,23 | p = 0,248 | R2 = 0,28 | p = 0,798 | AUC = 0,73 | p = 0,617 | |
| rs9818870 | ||||||||
| CC | N = 1.843 | 0,680 ± 0,003 | N = 1.843 | 0,867 ± 0,005 | N = 1.864 | 5,977 ± 0,050 | N = 1.899 | 1 |
| CT | N = 582 | 0,674 ± 0,005 | N = 582 | 0,864 ± 0,009 | N = 591 | 6,005 ± 0,088 | N = 605 | 1,06 [0,64-1,74] |
| TT | N = 35 | 0,648 ± 0,022 | N = 35 | 0,820 ± 0,038 | N = 35 | 5,813 ± 0,362 | N = 36 | 2,45 [0,70-8,55] |
| R2 = 0,33 | p = 0,252 | R2 = 0,23 | p = 0,460 | R2 = 0,27 | p = 0,864 | AUC = 0,73 | p = 0,414 | |
| rs12526453 | ||||||||
| CC | N = 1.013 | 0,675 ± 0,004 | N = 1.013 | 0,864 ± 0,007 | N = 1.028 | 5,958 ± 0,067 | N = 1.052 | 1 |
| CG | N = 1.136 | 0,678 ± 0,004 | N = 1.136 | 0,867 ± 0,007 | N = 1.141 | 5,941 ± 0,063 | N = 1.163 | 0,85 [0,55-1,33] |
| GG | N = 314 | 0,681 ± 0,007 | N = 314 | 0,862 ± 0,013 | N = 323 | 6,147 ± 0,119 | N = 326 | 0,55 [0,26-1,20] |
| R2 = 0,33 | p = 0,730 | R2 = 0,23 | p = 0,926 | R2 = 0,28 | p = 0,298 | AUC = 0,74 | p = 0,228 | |
| rs1333049 | ||||||||
| CC | N = 654 | 0,685 ± 0,005 | N = 654 | 0,870 ± 0,009 | N = 661 | 6,005 ± 0,084 | N = 674 | 1 |
| GC | N = 1.230 | 0,680 ± 0,004 | N = 1.230 | 0,870 ± 0,007 | N = 1.253 | 5,982 ± 0,061 | N = 1.272 | 0,80 [0,49-1,31] |
| GG | N = 599 | 0,668 ± 0,005 | N = 599 | 0,857 ± 0,009 | N = 601 | 5,947 ± 0,088 | N = 615 | 0,69 [0,38-1,26] |
| R2 = 0,33 | p = 0,065 | R2 = 0,23 | p = 0,488 | R2 = 0,27 | p = 0,890 | AUC = 0,74 | p = 0,473 | |
| rs1746048 | ||||||||
| CC | N = 1.864 | 0,676 ± 0,003 | N = 1.864 | 0,864 ± 0,005 | N = 1.883 | 5,963 ± 0,050 | N = 1.919 | 1 |
| TC | N = 565 | 0,687 ± 0,006 | N = 565 | 0,880 ± 0,010 | N = 576 | 6,042 ± 0,090 | N = 585 | 1,30 [0,81-2,09] |
| TT | N = 49 | 0,642 ± 0,019 | N = 49 | 0,802 ± 0,033 | N = 50 | 5,687 ± 0,305 | N = 51 | 0,66 [0,09-4,89] |
| R2 = 0,33 | p = 0,044 | R2 = 0,23 | p = 0,046 | R2 = 0,27 | p = 0,472 | AUC = 0,74 | p = 0,507 | |
| rs9982601 | ||||||||
| CC | N = 1.876 | 0,676 ± 0,003 | N = 1.876 | 0,865 ± 0,005 | N = 1.896 | 6,031 ± 0,049 | N = 1.941 | 1 |
| CT | N = 551 | 0,683 ± 0,006 | N = 551 | 0,867 ± 0,010 | N = 560 | 5,812 ± 0,090 | N = 565 | 0,92 [0,55-1,56] |
| TT | N = 32 | 0,682 ± 0,023 | N = 32 | 0,859 ± 0,040 | N = 34 | 5,487 ± 0,367 | N = 33 | 1,89 [0,43-8,40] |
| R2 = 0,33 | p = 0,619 | R2 = 0,23 | p = 0,970 | R2 = 0,28 | p = 0,043 | AUC = 0,74 | p = 0,729 | |
| rs10455872 | ||||||||
| AA | N = 2.121 | 0,679 ± 0,003 | N = 2.121 | 0,867 ± 0,005 | N = 2.143 | 5,964 ± 0,047 | N = 2.189 | 1 |
| GA | N = 370 | 0,683 ± 0,007 | N = 370 | 0,870 ± 0,012 | N = 381 | 6,031 ± 0,111 | N = 389 | 1,96 [1,21-3,16] |
| GG | N = 13 | 0,670 ± 0,037 | N = 13 | 0,911 ± 0,064 | N = 14 | 5,660 ± 0,581 | N = 14 | — |
| R2 = 0,33 | p = 0,799 | R2 = 0,23 | p = 0,769 | R2 = 0,27 | p = 0,741 | AUC = 0,75 | p = 0,022 | |
| ALOX5AP haplotipo B | ||||||||
| 0* | N = 1.433 | 0,679 ± 0,003 | N = 1.433 | 0,870 ± 0,006 | N = 1.452 | 5,990 ± 0,057 | N = 1.434 | 1 |
| 1* | N = 902 | 0,679 ± 0,004 | N = 902 | 0,865 ± 0,008 | N = 916 | 5,960 ± 0,071 | N = 894 | 0,86 [0,55-1,34] |
| 2* | N = 159 | 0,669 ± 0,010 | N = 159 | 0,853 ± 0,018 | N = 160 | 6,010 ± 0,171 | N = 157 | 0,49 [0,15-1,60] |
| R2 = 0,33 | p = 0,629 | R2 = 0,23 | p = 0,655 | R2 = 0,27 | p = 0,930 | AUC = 0,75 | p = 0,381 | |
AUC: área bajo la curva; GIMacc: grosor intimomedial de la arteria carótida común; ITB: índice tobillo-brazo.
Se muestra media ± error estándar de la media y los máximos grosor intimomedial de la arteria carótida común y rigidez carotídea; odds ratio [intervalo de confianza del 95%] de los individuos con índice tobillo-brazo patológico. Se muestra la variabilidad (R2) o la capacidad de discriminación (área bajo la curva) explicada con cada modelo.
Coeficientes de regresión con ajuste por edad y sexo para la asociación entre la puntuación de riesgo genético (por cada 1 desviación estándar) y diferentes medidas de la aterosclerosis subclínica
| sGRS | p | R2/AUC | |
|---|---|---|---|
| GIMbul máximo | 0,006 ± 0,009 | 0,488 | 0,16 |
| GIMacc máximo | 0,001 ± 0,005 | 0,792 | 0,23 |
| GIMaci máximo | 0,005 ± 0,006 | 0,423 | 0,08 |
| GIMbul medio | 0,006 ± 0,005 | 0,191 | 0,19 |
| GIMacc medio | 0,003 ± 0,003 | 0,370 | 0,33 |
| GIMaci medio | 0,004 ± 0,004 | 0,253 | 0,09 |
| Rigidez media | 0,000 ± 0,045 | 0,997 | 0,27 |
| ITB patológico | –0,030 ± 0,111 | 0,793 | 0,74 |
AUC: área bajo la curva; GIMacc: grosor intimomedial de la arteria carótida común; GIMaci: grosor intimomedial de la arteria carótida interna; GIMbul: grosor intimomedial en el bulbo carotídeo; ITB: índice tobillo-brazo; sGRS: puntuación de riesgo genético estandarizada.
Coeficientes lineales de las mediciones carotídeas ± error estándar, valor de p y variabilidad explicada por el modelo (R2) y coeficiente logístico (error estándar; valor de p y área bajo la curva receiver operating characteristic del modelo) para el índice tobillo-brazo patológico.
El método de evaluación de la rigidez carotídea o arterial fue muy heterogéneo entre los distintos estudios, y el número de estudios que evaluaron este fenotipo es escaso; en consecuencia, en el metanálisis no se tuvo en cuenta este marcador de la aterosclerosis. Murabito et al. han publicado recientemente un metanálisis de estudios del ITB que incluye a más de 60.000 participantes27. Por consiguiente, se limitó el metanálisis al GIMacc.
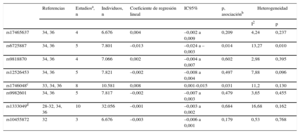
El proceso utilizado para la selección de los estudios incluidos en el metanálisis se resume en la tabla 2 del material suplementario. Finalmente se incluyeron nueve artículos28–36, y el número de participantes incluidos en el metanálisis fue de entre 6.676 (rs17465637 y rs10455872) y 32.056 (rs1333049). Los resultados presentados en los estudios incluidos en el metanálisis se muestran en la tabla 3 del material suplementario. Los resultados del metanálisis indican asociación negativa entre el alelo de riesgo rs6725887 (C) y el GIM carotídeo y asociación positiva entre el alelo de riesgo rs1746048 (C) y el GIM carotídeo (tabla 6). Había heterogeneidad entre los estudios que evaluaron la asociación entre rs6725887 y el GIM carotídeo (p = 0,010) (tabla 6). En el análisis de metarregresión se identificó que el GIM medio de la población explicaba esa heterogeneidad: había asociación en las poblaciones con GIM medio más alto, pero no se observaba en cambio en las que tenían un GIM más bajo (figura del material suplementario). No se observaron asociaciones entre las demás variantes genéticas seleccionadas y el GIMacc (tabla 6). Se dispuso de una potencia estadística del 80% para la detección de un coeficiente β situado entre > 0,007mm para rs1333049 y > 0,027mm para rs10455872. Los gráficos de embudo no indican que haya sesgo de publicación.
Resultados del metanálisis de la asociación entre las variantes genéticas de interés y el grosor intimomedial de la arteria carótida común
| Referencias | Estudiosa, n | Individuos, n | Coeficiente de regresión lineal | IC95% | p, asociaciónb | Heterogeneidad | ||
|---|---|---|---|---|---|---|---|---|
| I2 | p | |||||||
| rs17465637 | 34, 36 | 4 | 6.676 | 0,004 | –0,002 a 0,009 | 0,209 | 4,24 | 0,237 |
| rs6725887 | 34, 36 | 5 | 7.801 | –0,013 | –0,024 a –0,003 | 0,014 | 13,27 | 0,010 |
| rs9818870 | 34, 36 | 4 | 7.066 | 0,002 | –0,004 a 0,007 | 0,602 | 2,98 | 0,395 |
| rs12526453 | 34, 36 | 5 | 7.821 | –0,002 | –0,008 a 0,004 | 0,497 | 7,88 | 0,096 |
| rs1746048c | 33, 34, 36 | 8 | 10.581 | 0,008 | 0,001-0,015 | 0,031 | 11,2 | 0,130 |
| rs9982601 | 34, 36 | 5 | 7.817 | –0,002 | –0,007 a 0,003 | 0,479 | 3,65 | 0,455 |
| rs1333049d | 28-32, 34, 36 | 10 | 32.056 | –0,001 | –0,003 a 0,002 | 0,684 | 16,68 | 0,162 |
| rs10455872 | 32 | 3 | 6.676 | –0,003 | –0,006 a 0,001 | 0,179 | 0,53 | 0,768 |
IC95%: intervalo de confianza del 95%.
En este estudio se presenta la ausencia de asociación entre nueve variantes genéticas asociadas a la EC pero no a los FRCVc y tres mediciones de la aterosclerosis subclínica (GIM carotídeo, rigidez carotídea e ITB) en una encuesta de base poblacional. Este resultado se observa tanto con las variantes analizadas individualmente como considerando la puntuación de riesgo genético ponderada. Además, se ha resumido toda la información disponible en diferentes metanálisis y se ha observado que solo el alelo de riesgo rs1746048 (CXCL12) tiene asociación directa con el GIM carotídeo, mientras que el alelo de riesgo rs6725887 (WDR12) muestra asociación inversa con el GIM carotídeo (contrariamente a lo esperado).
Estos resultados amplían lo observado en tres estudios recientes con objetivos similares. Conde et al35 utilizaron datos de tres estudios europeos de base poblacional para evaluar la asociación entre 12 variantes genéticas (siete de ellas incluidas en el presente estudio, que añadían el SNP LPA y el ALOX5AP HapB) y el GIM medio de la arteria carótida común; ninguna de las variantes individuales se asoció al parámetro de interés. Hernesniemi et al36 analizaron la asociación entre una puntuación de riesgo genético basada en 24 SNP identificados en un estudio de genoma completo como asociados a la EC (siete de ellos incluidos en nuestro estudio) y el GIM carotídeo y la elasticidad carotídea en dos poblaciones finlandesas y una estadounidense. Estos autores no observaron asociación alguna con el GIM carotídeo determinado en dos momentos diferentes (2001 y 2007) ni con la progresión del grosor en ese periodo de 6 años. También analizaron la asociación de los diversos SNP con las mediciones de la aterosclerosis subclínica; aunque dos de los SNP tendían a presentar asociación con el GIM carotídeo (rs4977574) o la elasticidad carotídea (rs4773144), no se pudo reproducir estos resultados36. Por último, Zhang et al28 seleccionaron 10 SNP asociados con la EC (solo rs1333049 en común con el presente estudio) y analizaron su asociación con cuatro mediciones de la aterosclerosis subclínica (GIMacc y GIMaci, presencia de placa carotídea e ITB patológico) en cinco encuestas de base poblacional. Estos autores observaron que rs780094 (GCKR) se asocia a placa carotídea en la población amerindia estadounidense, pero no en los estadounidenses de origen europeo, afroamericano o mexicano. No se registró ninguna otra asociación entre los demás SNP y los demás fenotipos intermedios de interés28.
Los resultados del metanálisis respaldan la ausencia de asociación entre la mayor parte de estas variantes genéticas y la aterosclerosis subclínica. Estos resultados tienen varias explicaciones posibles.
En primer lugar, los mecanismos que explican la asociación entre estas nueve variantes genéticas y la EC no están relacionados con el remodelado arterial, la esclerosis o una limitación del flujo distal, y probablemente no se manifiestan en la pared arterial y no se ven reflejadas en cambios del GIM carotídeo, la rigidez o el ITB. Otros marcadores de la aterosclerosis subclínica, como la presencia de placa, podrían ser más sensibles para detectar esta progresión; algunos estudios han puesto de manifiesto el importante papel predictivo de la presencia y el grado de placas ateroscleróticas determinados mediante tomografía computarizada como posible sustrato para los eventos coronarios37. Una explicación más específica puede estar relacionada con la estabilidad de la placa; algunas variantes genéticas podrían predisponer al desarrollo de placas de aterosclerosis estables, mientras que otras podrían predisponer a la inestabilidad de la placa, con el consiguiente aumento del ulterior riesgo de rotura, trombosis e infarto agudo de miocardio. Un reciente estudio ha demostrado que hay variantes genéticas específicas y diferentes que fomentan el desarrollo de la aterosclerosis coronaria (en el gen ADAMTS7), mientras que otras se asocian a eventos agudos, como el infarto de miocardio (en el gen ABO)38. A favor de este argumento, se observa que solo el alelo de riesgo rs1746048 del gen CXCL12 se asocia al GIM carotídeo en el sentido esperado. La proteína CXCL12 se ha asociado a estabilización de la placa39 y podría facilitar el remodelado arterial que puede detectarse por un engrosamiento de la pared de la arteria. En cambio, el alelo de riesgo rs6725887 del gen WDR12 se asocia a mayor riesgo de EC pero con menor GIM carotídeo. El gen WDR12 codifica una proteína que interviene en diversos procesos celulares como la progresión del ciclo celular y la proliferación celular. Este alelo de riesgo podría inhibir la proliferación de las células de músculo liso, comprometer la estabilidad de la placa y aumentar el riesgo de eventos agudos sin inducir un engrosamiento de la arteria.
Otra posible explicación podría estar relacionada con mecanismos diferentes y progresión de la aterosclerosis en distintos territorios arteriales. Bis et al40 identificaron tres variantes genéticas asociadas al GIM carotídeo y dos asociadas a la placa carotídea en un metanálisis de nueve poblaciones participantes en el consorcio CHARGE. La única variante asociada a la aterosclerosis carotídea en ese metanálisis y también a la EC en el consorcio CARDIoGRAM fue la de rs1878406 (EDNRA). Por otro lado, tan solo hay un estudio de genoma completo que haya tenido como objetivo identificar variantes genéticas asociadas al ITB, que incluyó 21 estudios de base poblacional27. Solo se identificó una asociación de genoma completo significativa con el ITB, en el cromosoma 9p21 (rs10757269 en LD con rs1333049; R2 = 0,87). Aunque este locus se asocia también a la EC, las demás variantes asociadas a la EC no se asociaban al ITB.
Este estudio tiene algunas posibles limitaciones. En primer lugar, el tamaño muestral es pequeño y no se puede descartar que la magnitud de la asociación sea demasiado baja para detectarla con esta potencia estadística; no obstante, el metanálisis de toda la información disponible se diseñó para aumentar la potencia estadística del estudio. En segundo lugar, solo se pudo incluir en el estudio a los participantes que acudieron a la visita de seguimiento, y no se puede descartar la posibilidad de un sesgo de selección que pudiera afectar a los resultados, probablemente favorable a la hipótesis nula. En tercer lugar, no se incluyeron las variantes genéticas asociadas a la EC de manera independiente de los FRCVc que se han identificado más recientemente en el consorcio CARDIoGRAM-C4D. Entre los puntos fuertes del estudio, está el hecho de que se incluyeran varias mediciones subclínicas obtenidas con métodos estandarizados en la población de estudio y que se analizara el efecto individual y conjunto de las variantes genéticas seleccionadas.
CONCLUSIONESLos resultados indican que las variantes genéticas analizadas que se asocian a la EC de manera independiente de los FRCVc influyen en el riesgo de EC por mecanismos independientes de la aterosclerosis subclínica medida con el GIM carotídeo, la rigidez carotídea o el ITB, con la excepción de rs1746048 (CXCL12) y rs6725887 (WDR12). Estos resultados generan hipótesis respecto a que la variante CXCL12 parece influir en el riesgo de EC a través del remodelado y el engrosamiento de las arterias, mientras que la variante de riesgo WDR12 podría estar relacionada con una mayor vulnerabilidad de la placa. Serán necesarios estudios funcionales para confirmar tales hipótesis.
FINANCIACIÓNEsta investigación tuvo el respaldo de la financiación del Gobierno de España a través del Ministerio de Ciencia e Innovación, Instituto de Salud Carlos III, Fondo de Desarrollo Regional Europeo (Red HERACLES RD12/0042); Fondo de Investigación Sanitaria (FIS PI11/01801; PS09/00456); CIBERESP; Fundació La Marató de TV3 (081632), y Agència de Gestió d’Ajuts Universitaris i de Recerca (2009 SGR 1195).
CONFLICTO DE INTERESESNinguno.
Los autores agradecen a Susanna Tello, Marta Cabañero y Leny Franco la gestión de proyecto y de los datos. Agradecemos también la corrección del texto inglés por parte de Elaine Lilly de Writer's First Aid.