Los microARN (miARN) son una clase de ARN no codificante de pequeño tamaño (20-25 nucleótidos) que participan en la regulación génica. En los últimos años, los miARN han emergido como un mecanismo epigenético clave en el desarrollo y en la funcionalidad del sistema cardiovascular. Estas especies moleculares regulan funciones básicas en prácticamente todos los tipos celulares y por ello están directamente asociadas con la fisiopatología de un gran número de enfermedades cardiovasculares. Desde su relativamente reciente descubrimiento en fluidos extracelulares, se ha estudiado los miARN como potenciales biomarcadores de enfermedad. Son numerosos los estudios que proponen a los miARN como biomarcadores circulantes de diferentes patologías cardiovasculares (infarto de miocardio, enfermedad coronaria o insuficiencia cardiaca, entre otras), incluso con propiedades fisicoquímicas y bioquímicas superiores a los indicadores proteicos convencionales utilizados actualmente en la práctica clínica. En el presente trabajo se ofrece una breve introducción al campo de los miARN, con especial atención a su potencial aplicación clínica. Esta incluye su posible papel como nuevos biomarcadores de diagnóstico o de pronóstico en la enfermedad cardiovascular.
Palabras clave
Tan solo un 1-3% del genoma humano corresponde a secuencias de nucleótidos que codifican proteínas1. El 97-99% restante se consideraba originalmente como ADN basura (junk) ya que se hipotetizó que estas secuencias no codificaban información biológicamente relevante. El número de genes que codifican proteínas es similar entre especies con alto y bajo grado de complejidad biológica. Por tanto, el grado de complejidad biológica no puede explicarse únicamente por el flujo unidireccional gen-proteína2. La complejidad biológica podría estar determinada por la regulación de la expresión génica3. En la actualidad, se considera que los ARN no codificantes (ARNnc), ARN no traducidos a proteína claves en la regulación de la expresión génica, son una de las principales fuentes de esta complejidad4. En efecto, la proporción de genes que no codifican información para proteínas frente a genes que codifican información proteica es 17 veces mayor en los seres humanos en comparación con el nematodo Caenorhabditis elegans5.
Los ARNnc se clasifican en 2 subclases: ARNnc de cadena larga (mayores de 200 nucleótidos) y ARNnc pequeños (menores de 200 nucleótidos). Los microARN (miARN), con un tamaño de 19-25 nucleótidos, son el grupo de ARN de pequeño tamaño que ha atraído mayor atención durante los últimos años. El primer miARN fue descubierto en 1993 en C. elegans6,7. Hasta principios del siglo xxi no se describieron los primeros miARN en humanos8. Desde entonces, este campo de investigación ha crecido de forma exponencial. Al igual que otros mecanismos epigenéticos previamente descritos, como la metilación del ADN o la modificación de histonas, los miARN son importantes reguladores de la expresión génica. Son moléculas muy conservadas y presentes en todos los tipos celulares. Participan en la mayoría (si no en todos) de los procesos biológicos9 constituyendo un mecanismo de control epigenético fundamental a nivel postranscripcional que permite la regulación temporal de la expresión del ARN mensajero (ARNm) y, por ello, la rápida respuesta a los cambios ambientales10. Se han identificado alrededor de 2.500 miARN humanos, catalogados en la base de datos miRBase11, aunque en trabajos recientes se ha propuesto la existencia de un repertorio todavía mayor12.
El presente artículo pretende proporcionar una breve descripción de la biología de los miARN intracelulares y extracelulares, así como resumir la información más reciente sobre la investigación de estas especies moleculares en la enfermedad cardiovascular, centrándose principalmente en su potencial como biomarcadores de futura aplicación clínica.
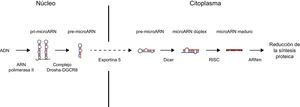
BIOGÉNESIS Y FUNCIÓN DE LOS MICROARNDurante su biogénesis, los miARN siguen un proceso de varios pasos que incluye: transcripción, maduración nuclear, exportación al citoplasma y posterior procesamiento (figura).
Biogénesis de microARN (miARN). En el núcleo, una gran proporción de genes de miARN se transcribe mediante la ARN polimerasa II (un grupo menor de estos mediante la ARN polimerasa III). La transcripción genera un miARN primario (pri-miARN), de cientos o miles de nucleótidos de longitud. En la vía de la biogénesis canónica miARN, el complejo de procesamiento de miARN (compuesto por la RNasa Drosha y el cofactor DGCR8) corta el pri-miARN generando el miARN precursor (pre-miARN) de 70 a 100 nucleótidos de longitud. Este pre-miARN se exporta desde el núcleo al citoplasma a través de la exportina 5. Una vez en el citoplasma, la enzima Dicer procesa y corta el pre-miARN generando un dúplex de pequeño tamaño (alrededor de 22 nucleótidos) que contiene el miARN maduro y la cadena complementaria. Cada dúplex contiene 2 miARN maduros: la hebra guía, con una prevalencia de 96-99%, y la hebra pasajera. Este dúplex se asocia con un miembro de la familia de proteínas de unión al ARN (AGO [argonauta]) que ensambla el complejo de silenciamiento inducido por ARN (RISC, del inglés RNA-induced silencing complex). La hebra guía se retiene en el complejo, mientras que la hebra pasajera se degrada. En algunos casos, ambas cadenas pueden ser funcionales. No todos los miARN conocidos se ajustan a esta ruta canónica. En una ruta alternativa los miARN (denominados mirtrones) derivan de los intrones generados durante el splicing del ARN mensajero (ARNm). La unión del complejo RISC-miARN al ARNm está mediada por un emparejamiento de Watson-Crick de la región seed del miARN, una secuencia de 6 a 8 nucleótidos en el extremo 5’ del miARN, con secuencias complementarias generalmente en la región no traducida 3’ (UTR) del ARNm diana. También se han descrito interacciones con las regiones codificantes y 5’ UTR del ARNm. Una vez unido, el RISC es capaz de inducir el silenciamiento de la expresión génica a nivel postranscripcional mediante la degradación o la inhibición de la traducción del ARNm; lo que conduce en ambos casos a la reducción de los niveles de proteína diana correspondiente. El mecanismo predominante por el cual los miARN reducen la síntesis de proteínas es la deadenilación del ARNm diana, fenómeno que aumenta la susceptibilidad del ARNm a la degradación.
En cuanto a su función, los miARN actúan a nivel postranscripcional mediante la degradación del ARNm o la inhibición de la traducción y en ambos casos disminuyen la expresión del gen sobre cuyo ARNm hacen diana. Generalmente solo existe complementariedad parcial entre los nucleótidos de la región seed del miARN (una secuencia de 6 a 8 nucleótidos en el extremo 5’) y la del ARNm diana. La complementariedad perfecta entre miARN y ARNm no es fundamental para reducir la expresión génica y, de hecho, rara vez ocurre en mamíferos. Tanto el pequeño tamaño de la región seed como el incompleto emparejamiento con el ARNm diana permiten que un mismo miARN pueda suprimir la expresión de diferentes genes, a menudo funcionalmente relacionados. A su vez, un mismo ARNm puede contener múltiples sitios de unión para varios miARN y generar una compleja red de interacciones miARN-ARNm. De esta forma, se estima que los miARN tienen la capacidad de regular la expresión de aproximadamente el 60% de los genes humanos13. Así pues, los miARN constituyen una densa red reguladora de la expresión génica con múltiples efectos cooperativos sobre un gran número de genes; lo que permite controlar diversas rutas moleculares a distintos niveles2.
MICROARN EXTRACELULARES: NUEVOS MEDIADORES EN LA COMUNICACIÓN INTERCELULARPese a que el primer trabajo que describió la presencia de ARN extracelular intacto en la circulación sanguínea se publicó en el año 197214, hasta el año 2007 no se propuso la existencia de miARN extracelulares15. Un año después, en un trabajo clave para este campo, Mitchell et al.16 demostraron la existencia de miARN en la circulación sanguínea. Desde entonces se ha descrito la presencia de miARN en un amplio número de fluidos corporales17.
Al contrario que otros tipos de ARN como el ARNm, los miARN son altamente estables en el medio extracelular. Los miARN extracelulares están protegidos de la degradación por el empaquetamiento en diferentes tipos de vesículas lipídicas18, tales como cuerpos apoptóticos, micropartículas y exosomas, y por la unión con proteínas como AGO2 (argonauta 2) o NPM1 (nucleofosmina 1)19 o lipoproteínas, incluyendo lipoproteínas de alta y baja densidad20. La interacción con elementos lipídicos o proteicos confiere a los miARN una gran resistencia a diferentes condiciones adversas, como la degradación por la actividad de ribonucleasa, los pH extremos, las temperaturas elevadas, los ciclos repetidos de congelación-descongelación o el almacenamiento prolongado16. De hecho, los miARN sintéticos libres se degradan rápidamente en el plasma sanguíneo16.
La incorporación de miARN en microvesículas ocurre de forma selectiva21. Nuestro grupo ha demostrado previamente que el perfil de miARN en microvesículas secretadas no se correlaciona necesariamente con el perfil intracelular de la célula de origen22. No obstante, los datos sobre la maquinaria celular implicada en el empaquetamiento de miARN en el interior de microvesículas o en la interacción proteína/lipoproteína-miARN son muy limitados20. Del mismo modo, aún se está lejos de comprender completamente los mecanismos que regulan la selección de miARN para exportar. En este sentido, Villarroya-Beltri et al.23 han descrito que la célula selecciona los miARN para exportar en exosomas mediante el reconocimiento de motivos específicos en la secuencia de nucleótidos. Por su parte, otros autores proponen que el contenido de miARN en exosomas se regula por el contenido global de miARN intracelular24.
Los miARN extracelulares actúan como un auténtico sistema de comunicación intercelular regulando la expresión génica y el fenotipo de las células receptoras. Al igual que sus formas intracelulares, los miARN extracelulares participan tanto en respuestas fisiológicas y adaptativas como en el inicio y en el desarrollo de estados patológicos, incluyendo las enfermedades cardiovasculares25,26. Algunos ejemplos muestran resultados convincentes sobre la relevancia de los miARN extracelulares en la homeostasis y en la patología del sistema cardiovascular. Zernecke et al.27 proponen que los cuerpos apoptóticos generados durante la aterosclerosis son capaces de transmitir señales paracrinas mediante miARN regulando la estabilidad de la placa de ateroma. Hergenreider et al.28 describen un mecanismo de comunicación ateroprotector entre las células endoteliales y las células vasculares musculares lisas mediado por vesículas extracelulares, miR-143 y miR-145. Shan et al.29 demuestran que miR-223 se secreta por células sanguíneas y se internaliza por células de la pared vascular, por lo que desempeña un papel relevante en la fisiología de las células vasculares musculares lisas (proliferación, migración y apoptosis). Por ello, proponen que miR-223 constituye una señal genética endocrina entre células sanguíneas y células vasculares.
MICROARN EN LA FISIOLOGÍA Y PATOFISIOLOGÍA CARDIOVASCULARLa alteración de los valores de expresión de miARN afecta directamente a la expresión de sus ARNm diana y, por lo tanto, los miARN son elementos potencialmente causales de la enfermedad. Desde la publicación del primer trabajo donde se demostró el papel de los miARN en el desarrollo de la leucemia crónica30, numerosos estudios han correlacionado modificaciones en los perfiles de expresión de miARN con el inicio y la evolución de diferentes patologías incluyendo el cáncer, las enfermedades metabólicas o los trastornos neurológicos31. La relevancia de la regulación de la expresión génica a través de los miARN en el desarrollo y en la homeostasis del sistema cardiovascular, así como la implicaciones de los miARN en la patología cardiovascular, están bien establecidas32. Se han publicado excelentes revisiones sobre el papel de los miARN en algunos aspectos relacionados con la funcionalidad y con diversas patologías del sistema cardiovascular33–35.
La alteración de los perfiles de expresión en los distintos tejidos que conforman el sistema cardiovascular se asocia con trastornos como la insuficiencia cardiaca, la aterosclerosis y las cardiomiopatías de diversa etiología36. Los miARN se expresan en cardiomiocitos, fibroblastos, células endoteliales y células vasculares musculares lisas; controlan prácticamente todos los aspectos de la biología del sistema cardiovascular, incluyendo el remodelado y la fibrosis cardiaca, la apoptosis, la inflamación, la proliferación, la angiogénesis y el metabolismo. A este respecto, diferentes estudios demuestran que la sobreexpresión o la represión de miARN específicos en modelos in vitro e in vivo incluyendo, entre otros, miR-1, miR-126, miR-133a o miR-208a es un mecanismo clave en la patología cardiovascular36,37. Un buen ejemplo del papel de los miARN en el desarrollo y en la fisiopatología cardiovascular proviene del modelo murino knockout condicional de la enzima Dicer en el corazón, implicada en la biogénesis de los miARN. Este modelo murino presenta miocardiopatía dilatada e insuficiencia cardiaca38, así como defectos cardiovasculares congénitos39.
MICROARN CIRCULANTES COMO BIOMARCADORES DE ENFERMEDAD CARDIOVASCULARLa identificación de sujetos en fases subclínicas de la enfermedad y/o alto riesgo cardiovascular tiene implicaciones cruciales para la salud de los pacientes, así como para la gestión eficiente de los recursos de los sistemas de salud, especialmente en el contexto actual de alta prevalencia de mortalidad y morbilidad asociadas a enfermedad cardiovascular. Por ello es fundamental el descubrimiento de nuevos biomarcadores y la mejora de las herramientas diagnósticas, particularmente de los algoritmos de predicción de riesgo40,41.
Los miARN circulantes presentan las propiedades biológicas y fisicoquímicas apropiadas para constituir biomarcadores útiles en la práctica clínica3: a) se pueden obtener mediante técnicas mínimamente invasivas en tipos de muestras utilizadas actualmente en los laboratorios clínicos; b) pueden originarse a partir de células necróticas o secretarse activamente a partir de células vivas; c) el perfil de miARN circulantes puede presentar una alta especificidad en función del tejido y de la enfermedad; d) el perfil de miARN circulantes se altera en situaciones de estrés celular y en condiciones patofisiológicas; e) los miARN son altamente estables y presentan una vida media larga dentro de la muestra; f) como ácidos nucleicos, los miARN ofrecen ventajas sobre los biomarcadores utilizados en la actualidad en la práctica clínica: los biomarcadores basados en péptidos pueden presentar distintas variantes de la misma molécula y están sujetos a modificaciones postraduccionales que dificultan su detección. Debido a su pequeño tamaño y a su composición química, los miARN son moléculas menos complejas que la mayoría de las moléculas biológicas del plasma; lo que facilita su determinación; g) se pueden cuantificar con una alta sensibilidad y especificidad de forma eficiente, rentable y relativamente rápida en los laboratorios clínicos actuales mediante reacción en cadena de la polimerasa en tiempo real, y h) pueden obtenerse perfiles globales en un solo experimento mediante reacción en cadena de la polimerasa en tiempo real o técnicas de relativo fácil acceso como la secuenciación o microarrays.
Todas estas características han llevado a distintos autores a proponer la aplicación clínica de miARN a corto o medio plazo42. Un test sanguíneo basado en miARN podría ser una potente herramienta no invasiva que proporcionase al clínico una valiosa información para el correcto manejo del paciente.
A continuación se abordará el papel de los miARN como biomarcadores en algunas de las entidades clínicas que se engloban bajo el término «enfermedad cardiovascular», si bien cubrir todas las patologías y todos los miARN propuestos como biomarcadores va más allá del alcance de este artículo.
Síndrome coronario agudoUno de los principales campos explorados en el estudio de los miARN como biomarcadores de enfermedad cardiovascular es su potencial diagnóstico en el síndrome coronario (tabla).
Selección de estudios sobre microARN circulantes y enfermedad cardiovascular
| Diseño experimental | Resultados destacados | Estudio |
|---|---|---|
| SCA | ||
| 71 pacientes ingresados en el departamento de cardiología | Valores elevados de miR-1 y miR-133a en pacientes de IAM, agina de pecho inestable y cardiomiopatía de tako-tsubo | Kuwabara et al.43 |
| 444 pacientes con SCA | Valores elevados de miR-1, miR-133a y miR-208b en pacientes de IAM en comparación con pacientes de angina inestable | Widera et al.44 |
| 216 pacientes con IAMCEST | Correlación de miR-133a con marcadores pronósticos, incluyendo tamaño del infarto | Eitel et al.45 |
| 510 pacientes de IAM remitidos para reperfusión mecánica primaria y 87 controles sanos | Valores elevados de miR-208b y miR-499 en paciente de AMI y casi indetectables en los controles sanos. Los pacientes con IAMCEST presentaron valores superiores a los pacientes sin IAMCEST; miARN detectables en el plasma 1 h después del inicio del dolor torácico | Devaux et al.46 |
| 332 pacientes con sospecha de SCA en el servicio de urgencias | Valores elevados de miR-1, miR-21, miR-146a, miR-208a y miR-499 en pacientes con SCA, incluso en pacientes con hscTnT inicialmente negativa o inicio de síntomas < 3 h; miR-1, miR-21 y miR-499 aumentaron el valor diagnóstico de hscTnT | Oerlemans et al.47 |
| 99 pacientes sin IAMCEST, 81 pacientes con EAC sin IAM y 99 controles sanos | Valores elevados de miR-1, miR-21 miR-133a, miR-423-5p y miR-499-5p pacientes sin IAMCEST afrente a control. Valores elevados de miR-21 y miR-499-5p en pacientes sin IAMCEST frente a EAC | Olivieri et al.48 |
| Estudio en 3 fases con 315 pacientes, incluyendo a sujetos con angina inestable, dolor torácico de origen no coronario y controles sanos | El perfil de miARN compuesto por miR-132, miR-150 y miR-186 mostró el mayor poder diagnóstico de angina inestable | Zeller et al.49 |
| 1.155 pacientes con dolor torácico agudo en el servicio de urgencias | Valores elevados de miR-208b, miR-499 y miR-320a en pacientes con IAM en comparación con aquellos con otros diagnósticos | Devaux et al.50 |
| EAC | ||
| 67 pacientes con EAC y 31 voluntarios sanos | Valores circulantes reducidos de miR-126, miR-17, miR-92a, miR-145 y miR-155 y elevados de miR-133a y miR-208a en pacientes con EAC | Fichtlscherer et al.51 |
| 195 sujetos sometidos a angiografía coronaria para la evaluación del dolor torácico | Valores reducidos de miR-145 en pacientes de EAC. Valores de miR-145 asociados a gravedad | Gao et al.52 |
| IC | ||
| 33 pacientes con IC aguda y 20 controles sanos | Valores elevados de miR-499 en pacientes con IC aguda | Corsten et al.53 |
| 50 pacientes con disnea y 39 controles sanos | Valores elevados de miR-423 en pacientes con IC | Tijsen et al.54 |
| 30 pacientes con IC crónica y 30 controles sanos | Valores elevados de miR-22, miR92b, miR-320a y miR-423 en pacientes con IC sistólica | Goren et al.55 |
| RCV | ||
| 820 sujetos del estudio Bruneck (población general) | miR-126, miR-197 y miR-223 asociados con la incidencia de IAM | Zampetaki et al.56 |
| 873 pacientes con EAC | miR-197 y miR-223 predictores de mortalidad | Schulte et al.57 |
| 1.112 pacientes con EAC (430 pacientes con SCA y 682 pacientes con angina de pecho estable) | Valores de miR-132, miR-140-3p y miR-210 mejoraron la discriminación de un modelo de predicción de mortalidad | Karakas et al.58 |
EAC: enfermedad arterial coronaria; hscTnT: troponina T cardiaca de alta sensibilidad; IAM: infarto agudo de miocardio; IAMCEST: infarto de miocardio con elevación del segmento ST; IC: insuficiencia cardiaca; miARN: microARN; RCV: riesgo cardiovascular; SCA: síndrome coronario agudo.
La amplia mayoría de los trabajos se centran en el estudio de los valores plasmáticos de miARN enriquecidos en el tejido miocárdico (miR-1, miR-133a/b, miR-208a y miR-499), también enriquecidos en el músculo esquelético; y por ello conocidos como myomiR. La concentración de estos miARN en el plasma aumenta poco después de la necrosis del miocardio con cinéticas similares a las observadas para los indicadores cardiacos convencionales: como la troponina T cardiaca de alta sensibilidad43,45,46. Los valores de miARN aumentan en los gradientes transcoronarios de los pacientes con síndrome coronario agudo, lo que sugiere que los miARN se liberan desde los cardiomiocitos necróticos59. En efecto, Cheng et al.60 identificaron en un metanálisis a miR-133a y miR-499 como potenciales biomarcadores de infarto de miocardio con aplicación clínica. Se han propuesto otros miARN (miR-21, miR-92, miR-126 o miR-132) como indicadores con una alta sensibilidad y especificidad, no solo de infarto de miocardio, sino también de angina inestable49,50,61,62. Cabe destacar que los miARN circulantes podrían añadir un valor diagnóstico a las troponinas cardiacas47. El potencial de los miARN circulantes como biomarcadores se ha puesto especialmente de manifiesto en los estudios que analizan su capacidad para mejorar el diagnóstico en pacientes con dolor torácico47. En este sentido, Zeller et al.49 proponen que la combinación de miR-132, miR-150 y miR-186 presenta un valor diagnóstico para angina inestable superior al de indicadores clásicos combinados como la troponina I cardiaca, el péptido natriurético tipo B y la proteína C reactiva.
La capacidad de los miARN circulantes para aportar información en el diagnóstico de angina inestable y estable63, infarto de miocardio y angina inestable44, e infarto agudo de miocardio sin elevación del segmento ST e insuficiencia cardiaca aguda48 también se ha destacado en diferentes trabajos.
Enfermedad coronariaProteínas plasmáticas como el fibrinógeno o la proteína C reactiva se han propuesto como biomarcadores de enfermedad coronaria64. Sin embargo, el valor diagnóstico de estos indicadores es limitado, ya que los modulan factores no necesariamente relacionados con la enfermedad cardiovascular64. Las técnicas de imagen disponibles no permiten detectar fases tempranas de la enfermedad. Por ello es fundamental el desarrollo de nuevos biomarcadores suficientemente robustos para identificar y caracterizar lesiones ateroscleróticas, especialmente aquellas inestables65.
Varios estudios han analizado la asociación de los miARN circulantes con la enfermedad coronaria (tabla). En un trabajo pionero, Fichtlscherer et al.51 propusieron los valores circulantes de miR-126, miR-17, miR-92a, miR-133a, miR-145, miR-155 y miR-208a como indicadores de la presencia de enfermedad coronaria. Algunos de estos miARN han sido validados posteriormente en estudios independientes66. El perfil de miARN circulantes parece estar asociado no solamente con la presencia de lesión coronaria, sino también con la gravedad de la enfermedad52,66,67. Los miARN también se han propuesto como potenciales biomarcadores de placas vulnerables62. No obstante, gran parte de estos resultados se han obtenido en estudios caso-control con pequeñas poblaciones.
Insuficiencia cardiacaDistintos estudios han demostrado la existencia de un perfil específico de miARN circulantes en pacientes con insuficiencia cardiaca (tabla). Marfella et al.68, tras evaluar un panel de 84 miARN previamente asociados con las alteraciones estructurales en el corazón, detectaron un perfil circulante de 24 miARN característicos de pacientes con insuficiencia cardiaca. Salvo excepciones69, diferentes estudios apuntan a miR-423 como un potencial biomarcador asociado a la insuficiencia cardiaca. En uno de los primeros trabajos en este campo, Tijsen et al.54 identificaron 6 miARN elevados en pacientes con insuficiencia cardiaca, entre los que miR-423 mostró una fuerte asociación con el diagnóstico clínico de la enfermedad. Otros trabajos posteriores han validado estos resultados55. Cabe destacar que en diferentes estudios se ha sugerido el potencial de los miARN como nuevos biomarcadores de insuficiencia cardiaca con fracción de eyección preservada70, disfunción diastólica71 e insuficiencia cardiaca aguda53.
Riesgo cardiovascularActualmente, la evaluación del riesgo cardiovascular está basada exclusivamente en factores de riesgo clásicos establecidos como la hipertensión, la dislipemia, la diabetes o el tabaquismo. Desafortunadamente, estos factores de riesgo tradicionales no explican completamente el riesgo de evento cardiovascular. Gran parte de los eventos ocurren en pacientes con un riesgo bajo o intermedio que presentan un número reducido de factores de riesgo cardiovascular clásicos65. Por el contrario, una gran parte de los individuos clasificados según estos factores como de alto riesgo no experimenta ningún episodio cardiovascular; ni siquiera a largo plazo72. Así pues, existe un interés clínico evidente en el desarrollo de nuevos biomarcadores no invasivos y de fácil acceso que permitan mejorar significativamente la capacidad predictiva de los algoritmos desarrollados hasta la fecha, más allá de los factores de riesgo tradicionales3.
Varios trabajos han analizado la capacidad de los miARN circulantes para predecir futuros eventos cardiovasculares tanto en la población general como en pacientes con enfermedad coronaria conocida. Zampetaki et al.56 demostraron por primera vez el potencial de los miARN para predecir eventos adversos. Demostraron, en una cohorte de 820 individuos con un seguimiento de 10 años, una asociación significativa de 3 miARN (miR-126, miR-197 y miR-223) con el riesgo de infarto de miocardio incluso tras ajustar por posibles factores de confusión. El potencial pronóstico de miR-197 y miR-223 como predictores de mortalidad cardiovascular se corroboró en una cohorte de 873 pacientes con enfermedad coronaria57. Por su parte, miR-132, miR-140 y miR-210 también se asociaron con muerte cardiovascular en una cohorte similar de 1.112 pacientes58. Recientemente, Bye et al.73 han propuesto que un panel de 5 miARN (miR-106a-5p, miR-424-5p, let-7g-5p, miR-144-3p y miR-660-5p) son capaces de mejorar la predicción del riesgo de infarto de miocardio en individuos sanos. Estos resultados ponen de manifiesto la prometedora aplicabilidad de los miARN como biomarcadores en la prevención primaria y secundaria de la enfermedad cardiovascular.
Otras patologías cardiovascularesEl estudio de miARN circulantes como biomarcadores de otras patologías cardiovasculares, más allá de las mencionadas, se ha abordado en un número minoritario de trabajos. En este sentido, a pesar de su gran incidencia, las investigaciones que han analizado la asociación entre miARN circulantes e ictus son escasas. El análisis de la expresión de miARN en sangre periférica de pacientes jóvenes con ictus isquémico reveló que los perfiles de miARN en sangre periférica pueden ser potenciales biomarcadores en el diagnóstico del ictus isquémico cerebral74. El perfil sanguíneo de miARN también puede discriminar entre accidente cerebrovascular hemorrágico e isquémico75. Otros autores han demostrado que los miARN séricos son indicadores con utilidad clínica en el caso del aneurisma aórtico abdominal76. El posible papel de los miARN como biomarcadores de ateroesclerosis periférica también se ha evaluado. Stather et al.77 identificaron un perfil específico de enfermedad arterial periférica que presentó un buen potencial diagnóstico y ofreció, además, información sobre posibles rutas moleculares implicadas en la patogénesis de la enfermedad. Recientemente se ha revisado en detalle el posible papel de los miARN circulantes como biomarcadores de diferentes trastornos cardiovasculares, incluyendo la fibrilación auricular, la miocarditis, el embolismo pulmonar, la cardiomiopatía de tako-tsubo, las complicaciones vasculares ligadas a la diabetes o la demencia vascular78,79.
LIMITACIONES EN LA APLICACIÓN CLÍNICA DE MICROARN CIRCULANTES COMO BIOMARCADORESA pesar de los prometedores resultados de los miARN circulantes como nuevos biomarcadores de enfermedad cardiovascular, los miARN aún no han entrado a formar parte de la práctica clínica; principalmente a causa de la falta de grandes estudios de cohortes y por las limitaciones técnicas de su aplicación9,37. Existe además una gran heterogeneidad entre los estudios, incluyendo diferencias entre los miARN y las enfermedades estudiadas; así como en el diseño experimental: recogida y tratamiento de las muestras y metodología empleada para el análisis de los miARN80. En este sentido, un grupo notable de los trabajos publicados no han comparado el potencial diagnóstico o pronóstico de los miARN circulantes con indicadores clínicos previamente establecidos y actualmente utilizados en la práctica clínica: un paso clave en el desarrollo de nuevos biomarcadores. Otro aspecto destacable es la inadecuada selección de controles, especialmente para factores de confusión como edad, sexo, medicación y comorbilidades81. La falta de especificidad de los miARN propuestos como biomarcadores también constituye una limitación. Por ejemplo, miR-126 se ha asociado fuertemente con enfermedad coronaria51. No obstante, este mismo miARN también está íntimamente ligado a la insuficiencia cardiaca82 y a la diabetes83; lo que pone en duda su utilidad clínica.
La implementación de diagnósticos basados en miARN en la rutina clínica también se ha visto obstaculizada por dificultades técnicas. Todavía no se han establecido métodos estandarizados para el aislamiento y la cuantificación de los miARN circulantes. Esta situación dificulta la comparación de estudios llevados a cabo por diferentes laboratorios. Asimismo, las actuales técnicas de detección de miARN no permiten un diagnóstico rápido; tan necesario, por ejemplo, en el caso del infarto agudo de miocardio.
OTROS BIOMARCADORES CIRCULANTES EPIGENÉTICOS DE ENFERMEDAD CARDIOVASCULARLos enfoques basados en ARNnc proporcionan nuevas oportunidades para desarrollar nuevos biomarcadores. Durante los últimos años, además de los miARN, son varios los trabajos que proponen el uso de ARNnc de cadena larga circulantes (los mayores de 200 nucleótidos) como biomarcadores sensibles y específicos no solo de enfermedad cardiovascular84,85, sino también de respuesta terapéutica86. La aplicación clínica de estas especies está más avanzada en el campo de la oncología, donde se han desarrollado ensayos para la detección del ARNnc de cadena larga PCA3 en orina: un biomarcador altamente específico de cáncer de próstata87.
Otros mecanismos epigenéticos, como la metilación del ADN o la modificación de histonas, podrían constituir una potente fuente de biomarcadores circulantes. No obstante, muy pocos estudios han valorado el papel de estos mecanismos epigenéticos como indicadores útiles en la práctica clínica. Cabe destacar que recientemente se ha revisado la asociación entre la metilación del ADN y la presencia de enfermedad cardiovascular88. En efecto, la hipometilación de la secuencia LINE-1 en células de sangre periférica o el tejido adiposo visceral está asociada con diabetes, obesidad, valores reducidos de colesterol unido a lipoproteínas de alta densidad y valores elevados de colesterol total e inflamación, incluso independientemente de otros factores de riesgo89,90.
PERSPECTIVASNo hace más de una década que se describieron los primeros miARN extracelulares. A pesar del relativamente corto intervalo de tiempo que ha transcurrido, hemos sido testigos de una gran evolución en su posible aplicación clínica como biomarcadores. La comunidad científica tiene grandes expectativas en la utilización clínica de los miARN circulantes como herramientas diagnósticas y pronosticas de la enfermedad cardiovascular. El hecho de que puedan detectarse fácilmente mediante la metodología empleada actualmente en los laboratorios clínicos, así como su asociación con la patología cardiovascular (en algunos casos, superior a la observada con indicadores convencionales) sugiere su potencial valor como biomarcadores. Sin embargo, se debe abordar varios aspectos antes de que los miARN se establezcan como nuevas herramientas clínicas. En los próximos años, la replicación de los actuales resultados en estudios independientes, multicéntricos y con grandes tamaños poblacionales constituirá un paso clave para dilucidar la aplicabilidad clínica real de los miARN circulantes.
FINANCIACIÓNEl presente trabajo ha recibido ayudas del Instituto de Salud Carlos III, cofinanciado con fondos FEDER (Fondo Europeo de Desarrollo Regional) (V. Llorente-Cortés: FIS PI14/01729), el Instituto de Salud Carlos III (CIBER Cardiovascular: CB16/11/00403), la Fundació La Marató de TV3 (V. Llorente-Cortes: 201521-10) y el Instituto de Salud Carlos III, Ministerio de Economía y Competitividad (E. Iglesias-Gutiérrez: DEP2012-39262). D. de Gonzalo-Calvo tiene un contrato Sara Borrell (CD14/00109) del Instituto de Salud Carlos III. D. de Gonzalo-Calvo y V. Llorente-Cortés son miembros de la red europea Cardiolinc.
CONFLICTO DE INTERESESNinguno.


![Biogénesis de microARN (miARN). En el núcleo, una gran proporción de genes de miARN se transcribe mediante la ARN polimerasa II (un grupo menor de estos mediante la ARN polimerasa III). La transcripción genera un miARN primario (pri-miARN), de cientos o miles de nucleótidos de longitud. En la vía de la biogénesis canónica miARN, el complejo de procesamiento de miARN (compuesto por la RNasa Drosha y el cofactor DGCR8) corta el pri-miARN generando el miARN precursor (pre-miARN) de 70 a 100 nucleótidos de longitud. Este pre-miARN se exporta desde el núcleo al citoplasma a través de la exportina 5. Una vez en el citoplasma, la enzima Dicer procesa y corta el pre-miARN generando un dúplex de pequeño tamaño (alrededor de 22 nucleótidos) que contiene el miARN maduro y la cadena complementaria. Cada dúplex contiene 2 miARN maduros: la hebra guía, con una prevalencia de 96-99%, y la hebra pasajera. Este dúplex se asocia con un miembro de la familia de proteínas de unión al ARN (AGO [argonauta]) que ensambla el complejo de silenciamiento inducido por ARN (RISC, del inglés RNA-induced silencing complex). La hebra guía se retiene en el complejo, mientras que la hebra pasajera se degrada. En algunos casos, ambas cadenas pueden ser funcionales. No todos los miARN conocidos se ajustan a esta ruta canónica. En una ruta alternativa los miARN (denominados mirtrones) derivan de los intrones generados durante el splicing del ARN mensajero (ARNm). La unión del complejo RISC-miARN al ARNm está mediada por un emparejamiento de Watson-Crick de la región seed del miARN, una secuencia de 6 a 8 nucleótidos en el extremo 5’ del miARN, con secuencias complementarias generalmente en la región no traducida 3’ (UTR) del ARNm diana. También se han descrito interacciones con las regiones codificantes y 5’ UTR del ARNm. Una vez unido, el RISC es capaz de inducir el silenciamiento de la expresión génica a nivel postranscripcional mediante la degradación o la inhibición de la traducción del ARNm; lo que conduce en ambos casos a la reducción de los niveles de proteína diana correspondiente. El mecanismo predominante por el cual los miARN reducen la síntesis de proteínas es la deadenilación del ARNm diana, fenómeno que aumenta la susceptibilidad del ARNm a la degradación. Biogénesis de microARN (miARN). En el núcleo, una gran proporción de genes de miARN se transcribe mediante la ARN polimerasa II (un grupo menor de estos mediante la ARN polimerasa III). La transcripción genera un miARN primario (pri-miARN), de cientos o miles de nucleótidos de longitud. En la vía de la biogénesis canónica miARN, el complejo de procesamiento de miARN (compuesto por la RNasa Drosha y el cofactor DGCR8) corta el pri-miARN generando el miARN precursor (pre-miARN) de 70 a 100 nucleótidos de longitud. Este pre-miARN se exporta desde el núcleo al citoplasma a través de la exportina 5. Una vez en el citoplasma, la enzima Dicer procesa y corta el pre-miARN generando un dúplex de pequeño tamaño (alrededor de 22 nucleótidos) que contiene el miARN maduro y la cadena complementaria. Cada dúplex contiene 2 miARN maduros: la hebra guía, con una prevalencia de 96-99%, y la hebra pasajera. Este dúplex se asocia con un miembro de la familia de proteínas de unión al ARN (AGO [argonauta]) que ensambla el complejo de silenciamiento inducido por ARN (RISC, del inglés RNA-induced silencing complex). La hebra guía se retiene en el complejo, mientras que la hebra pasajera se degrada. En algunos casos, ambas cadenas pueden ser funcionales. No todos los miARN conocidos se ajustan a esta ruta canónica. En una ruta alternativa los miARN (denominados mirtrones) derivan de los intrones generados durante el splicing del ARN mensajero (ARNm). La unión del complejo RISC-miARN al ARNm está mediada por un emparejamiento de Watson-Crick de la región seed del miARN, una secuencia de 6 a 8 nucleótidos en el extremo 5’ del miARN, con secuencias complementarias generalmente en la región no traducida 3’ (UTR) del ARNm diana. También se han descrito interacciones con las regiones codificantes y 5’ UTR del ARNm. Una vez unido, el RISC es capaz de inducir el silenciamiento de la expresión génica a nivel postranscripcional mediante la degradación o la inhibición de la traducción del ARNm; lo que conduce en ambos casos a la reducción de los niveles de proteína diana correspondiente. El mecanismo predominante por el cual los miARN reducen la síntesis de proteínas es la deadenilación del ARNm diana, fenómeno que aumenta la susceptibilidad del ARNm a la degradación.](https://static.elsevier.es/multimedia/03008932/0000007000000009/v1_201708280842/S0300893217301586/v1_201708280842/es/main.assets/thumbnail/gr1.jpeg?xkr=eyJpdiI6IjdaTGwxa1VjcHRrQjlMSi9QWncxVVE9PSIsInZhbHVlIjoiRVJONlNJWVlKQWovN25MbDFmbEhGRFlUeWFROTJNSXNxemlBZFdSK2s1OD0iLCJtYWMiOiIyZTFkMzA1MDljNDZhY2ZjMzc0Y2FjNGE3MWY4NDczN2MzMzBkNTY0YWQ3Nzc0OGUzMDU1MjYzMTE0ZDkzMTU3IiwidGFnIjoiIn0=)